S1000-Nanopore 空间全长转录组
产品简介
产品基于百创S1000亚细胞级空间转录组芯片与ONT测序平台相结合,利用ONT平台的超长读长与S1000较传统平台而言更长的Spatial Barcode(70bp),在后续的数据拆分无需依赖NGS的数据。除空间位置基因表达情况外还可以获得基因结构信息的空间位置。
技术流程与管理
通过空间转录组的切片、组织优化(摸索最*透化时间)、基因表达步骤后。使得每个mRNA分子带有Spatial Barcode,在建库测序时选择ONT平台进行上机测序。通过后续的配套分析软件完成从下机数据到基因表达矩阵的构建。
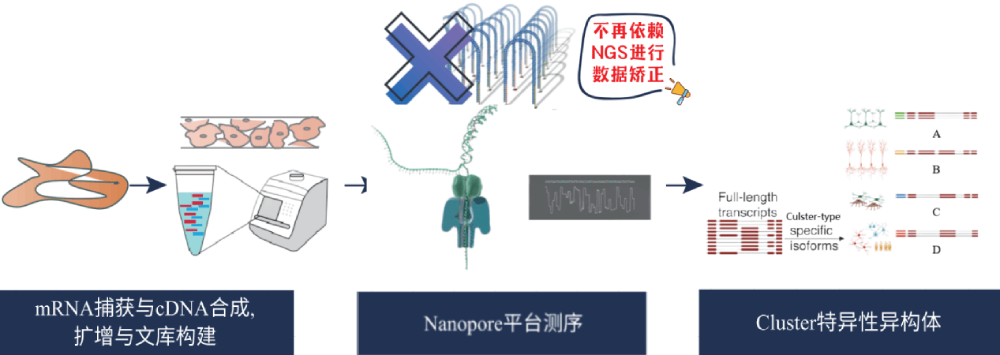
BMKMANU S1000 Full-length Transcriptome By Nanopore
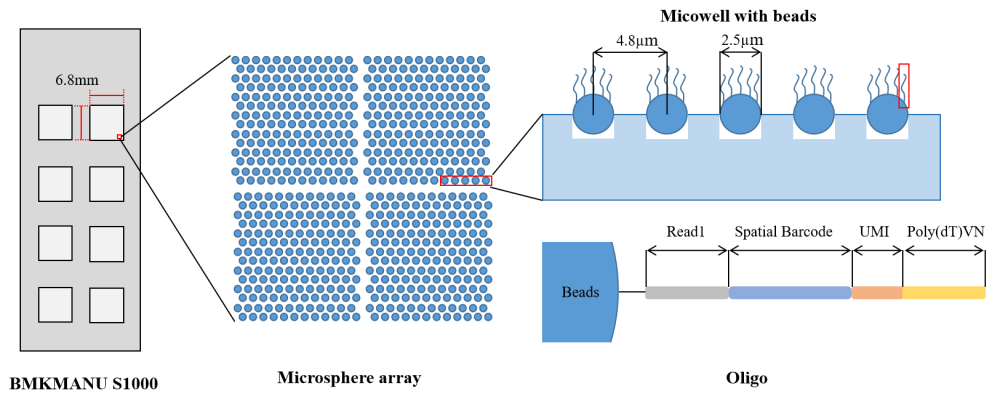
数据展示
TGS(Third-generation sequencing)数据
全长数据拆分出的Valid Barcode 百分比为73.85%,与二代数据(88.85%)相比,差别不大,相对于之前相差几倍而言这无疑是一个大的进步。数据比对到基因组,转录本,转录本区域内且拥有完整Barcode的比率分别为95.33%、66.44%、49.55%。
| Number of Reads | Number of Base | Reads with Valid Barcode | Bases with Valid Barcodes | N50 | Bases Mapped to Transcriptome | Bases Mappedto Transcriptome And with Valid Barcode |
| 105,553,898 | 70,516,281,320 | 73.85%(88.85%) | 73.42% | 740 | 66.44% | 49.55% |
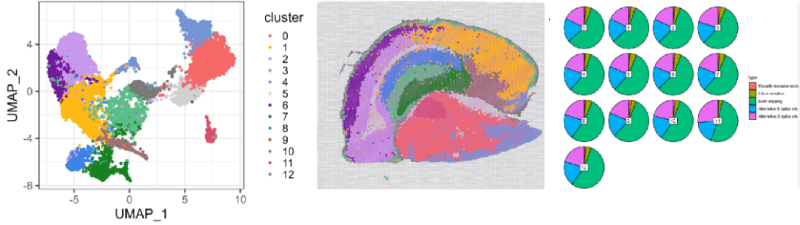
相关性分析与降维聚类
数据的相似性与准确性进行了展示与介绍,无论是二代与三代数据的UMI/Gene counts per spot的相关性还是tSEN/U-MAP的降维聚类,都显示该产品具有非常高的准确性。这充分说明百创S1000全长转录组产品完全可以独立进行数据的拆分与挖掘分析,无需依赖NGS数据的校正。
 可变剪接与基因融合
可变剪接与基因融合
空间三代全长转录组可以对结构进行分析。在50µm(Level7)分辨率下,对各Culster的可变剪接的事件数量进行了分析。结果显示外显子跳跃的类型最多。除可变剪接外,针对融合基因分析的结果同样进行了展示。
Level 7(50μm)下各Cluster中预测的可变剪接事件数量统计
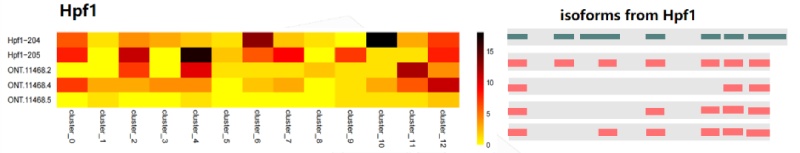
Hpf1 基因同源异构体表达热图及外显子使用
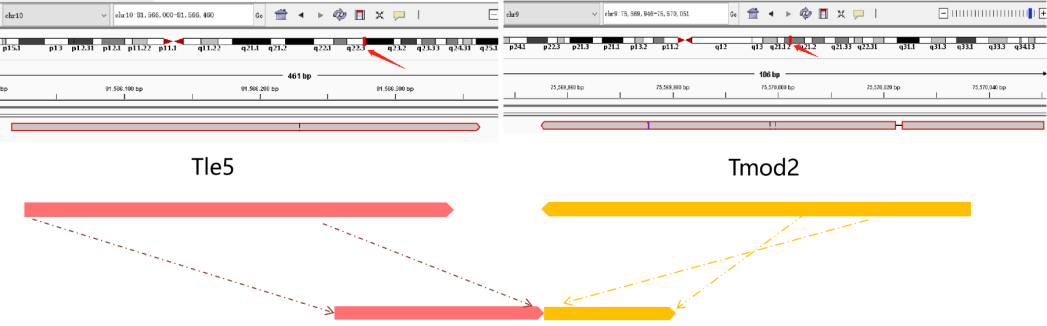
融合基因事件






 京公网安备 11011302003368号
京公网安备 11011302003368号